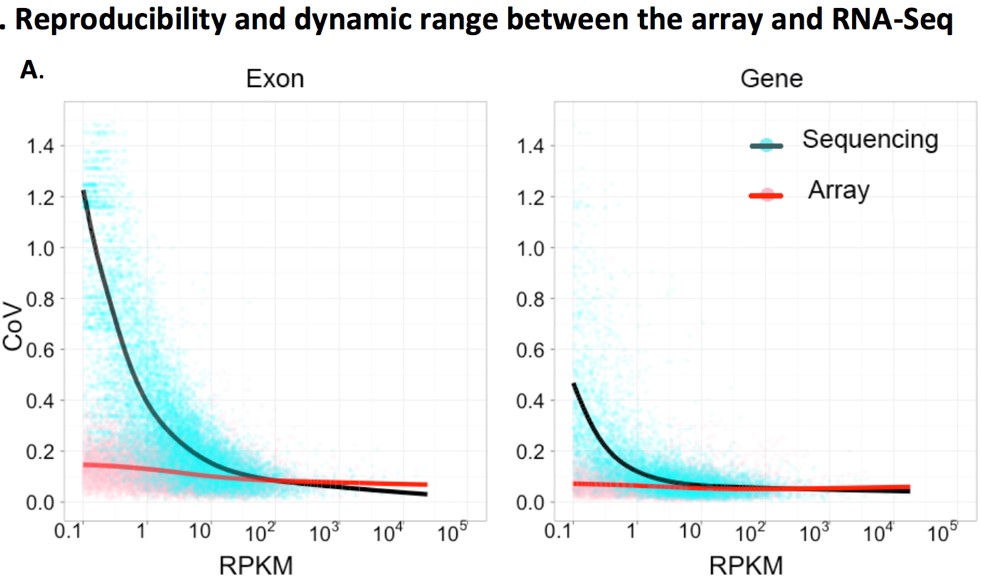
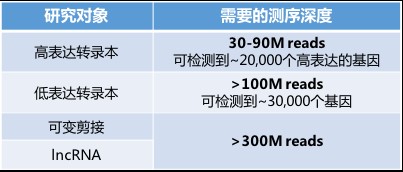
最近几年二代测序(又叫NGS)很火,而且价格越来越便宜,原来都用芯片检测mRNA、miRNA、LncRNA表达量的,好像不少都换用RNA-seq了。那么,到底选择哪种更好呢?今天就来回答下这个问题。一句话—— 看研究目的。 常见误区一: 对,但又不对。 首先,大家需要明确,检测到和准确分析基因表达量的概念是不同的,只有mapping到基因上的reads达到一定数量,才能得到相对准确的分析结果。因此RNA-Seq能检测到多少可靠的信息完全取决于测序深度,测序深度,测序深度!不同于芯片的杂交法,RNA-seq是通过读数来检测,读数多(即测序深度深)代表着RNA-seq的采样率高。采样率低了准确度自然就低了。 那么有没有一个实验能说明芯片和RNA-seq之间数据准确度的差异呢? 发表在PNAS上面的这篇文章就帮大家做了一个对比(PNAS 2011, 108(9):3707-3712.)。图中绿色点/黑色线是测序得到的数据,红色点/红色线是芯片得到的数据。在~50M reads数据量的情况下,当基因表达丰度较高时(横坐标RPKM较大时),两者之间的数据质量都是非常好的(纵坐标CoV即变异系数越小,数据质量越高),但当基因表达丰度变低时(横坐标RPKM较小时),RNA-seq的数据质量就急剧下降了,而芯片则仍然维持着高水准。这篇文章得到的结论是:~80%以上的基因,RNA-seq的数据质量/可信度都低于芯片。市场上最流行的的6G数据量的RNA-seq,其实就是40M reads或者20M paired reads,对于研究高表达丰度的基因来说,差不多是够用了。但是对于中、低表达丰度转录本就不够用了。 常见误区二: RNA-seq可以同时检测已知和未知基因,基因芯片只能检测已知基因,这是一个巨大的局限。 首先,这个观点的一个潜在假设是,每次测序都能够发现一些未知分子。但对于人、大鼠、小鼠以及其他一些模式生物,该发现的基因基本上都已经发现完了。因此基因是否已知,在很多情况下并非重点,重点在于该基因在您研究的领域中功能是否已知。芯片上已知基因的功能大多都还不清楚,只是盲目地去追求发现新分子并不可取。 在探索性研究和非模式生物研究中,RNA-seq才是更合适的选择。 常见误区三: RNA-seq现在已经很便宜了,比基因芯片还便宜很多。 测序中收费标准之一来源于数据量(即测序深度),刚刚说了,市场上最流行的的RNA-seq服务数据量是6G/样本,即40M reads或者20M paired reads ,这时候确实比很多芯片都便宜了。但是如果希望更准确检测中、低丰度RNA,就需要更深度的测序保证数据可靠性,这就会导致测序成本急剧上升。下表帮大家总结了一些常见研究的测序数据要求。Nature biotechnology有篇文章指出,如想要检测lncRNA、转录异构体等一般表达丰度极低的转录本,至少需要300M reads的测序量才能达到80%的数据准确度(Nature biotechnology, 2014, 32(9): 903-914.)。 那么芯片又如何呢?拿Affymetrix HTA系列的芯片来说,它的数据量,可是相当于480M reads测序深度!哇,好像看到了好多钱 🤑 常见误区四: RNA-seq在测表达量的同时还可以发现突变,基因芯片不能。 基因芯片(这里专指测RNA的表达谱芯片)确实不能发现突变。RNA-seq是通过测序来检测RNA丰度的,确实可以获得序列信息,但是因为测序本身有错误率,而RNA-seq常做的测序深度很低,得到的突变信息其实并不准确。要想准确,就需要极高的测序深度,那么又回到老问题了,成本基本是不可接受的。 那么有些同学要问啦,市场上的基因芯片有好多种啊,不知从何入手?小编下期再给大家聊聊基因芯片类型的选择。 长按加关注