2、mirbase数据库中human,mouse,rat全部成熟体序列microRNA;
3、circbase数据库中human,mouse全部circRNA;
其次,也是最重要的,就是基因坚持的三个最重要的设计原则:
1、引物跨内含子设计,避免基因组污染带来的影响;
2、引物在基因(NCBI Refseq)对应的所有转录本同源区设计;
3、引物特异性,设计引物全部使用NCBI primer-blast比对工具进行引物特异性比对;
最后,就是设计方法。qPCR使用的方法都是是染料法,使用的荧光染料就是常用的SYBRGreen I。该方法利用了SYBRGreen荧光染料非特异性与双链DNA结合并发光的特性,检测PCR的全部进程,从而判定初始RNA的量。染料法的优势是使用简单,成本也相对较低。下面简单介绍下mirbase数据库和circbase数据库中两类基因的qPCR引物设计方法。
1、circbase数据库中circRNA引物设计方法-常规qPCR设计方法
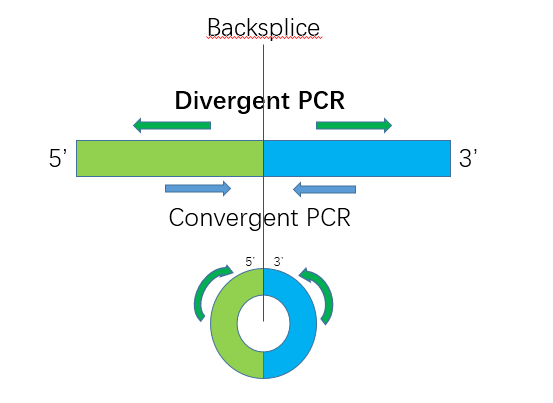
CircRNA成环方式有很多种,简单说明下外显子环化的circRNA和内含子环化的circRNA两类设计方法。对于外显子环化的circRNA,引物需要跨剪切位点(backsplice)设计;对于内含子环化circRNA,可跨剪切位点设计,也可围绕内含子区域设计引物。circRNA的正向引物和反向引物(Divergent PCR )与线性mRNA的正向引物和反向引物(convergent PCR)设计方向刚好是相反的。circRNA跨剪切位点(backsplice)设计示意图如下:
2、mirbase数据库中成熟体microRNA引物设计方法-茎环法
由于成熟体miRNA较小,无法用常规的方法直接进行反转录、PCR 。所以对于mirbase数据库中的成熟体microRNA使用的是茎环法,也就是将miRNA配对的序列延长,然后进行正常的反转录及后续的PCR检测。茎环法原理图如下:

引物产品具有高扩增效率和高灵敏度的特点,保证每次实验的可靠定量,无非特异性扩增和引物二聚体,确保实验的可重复性和实验数据的可靠性。
